近日,Journal of Agricultural and Food Chemistry在线发表了江苏大学食品与生物工程学院崔凤杰教授课题组的研究成果“Functional Characterization and Structural Basis of the β‑1,3-Glucan Synthase CMGLS from Mushroom Cordyceps militaris” (Fu et al., Journal of Agricultural and Food Chemistry, 2022, 70 (28), 8725-8737)。2020级硕士生付鑫为论文第一作者,崔凤杰教授为论文通讯作者。
葡聚糖是一大类由α/β-1,3、1,4或1,6-糖苷键连接葡萄糖单体形成的大分子多聚体,是真菌细胞壁的重要结构组分之一,其具有能量存储、细胞壁结构和信号传导等功能。β-葡聚糖因其重要的生物学功能而引起了广泛关注,例如刺激先天免疫、降低血脂、平衡肠道微生物菌群和促进伤口愈合。到目前为止,酵母和部分丝状真菌中β-葡聚糖的精细结构、生物学功能和合成途径已得到了较为详细的阐述,然而有关其合成相关酶系的催化特性和机制还未得到明确地阐明。
一般来说,β-葡聚糖的生物合成过程主要涉及前体UDP-葡萄糖的合成、β-葡聚糖链的聚合、伸长和分支,其关键步骤是由β-1,3-葡聚糖合成酶复合物通过催化亚基 β-1,3-葡聚糖合酶和至少一个调节亚基 Rho1在质膜(PM)附近发生的葡聚糖链的聚合和伸长。然而,β-1,3-葡聚糖合成酶复杂的空间结构严重阻碍了对其催化特性和机理的理解。
针对以上问题,该研究以蛹虫草为研究对象,来进一步明确蛹虫草中β-1,3-葡聚糖合成酶的催化特性和结构基础。首先,克隆、鉴定和重新注释了编码β-1,3-葡聚糖合酶的基因CMGLS,得到准确氨基酸序列后对其进行生物信息学分析。其次,采用产物包埋纯化法从蛹虫草发酵菌丝体中得到了部分纯化的CMGLS,并异源表达了葡聚糖合成酶的中心亲水结构域CMGLS-CHCD对比分析其催化特性。最后,通过AlphaFold首次预测了β-1,3-葡聚糖合成酶的三维(3D)结构(图1),并在此基础上,利用分子对接来阐明其潜在催化机制。结果表明底物UDP-葡萄糖通过氢键、π-π堆积、盐桥和疏水作用力与CMGLS中至少11个氨基酸残基结合,Arg1436被预测为直接与葡萄糖、磷酸和UDP-葡萄糖上的核糖环相互作用的关键氨基酸(图2)。这些发现将为认识和理解真菌葡聚糖合成酶催化合成葡聚糖的过程和机理提供了一定的研究基础和技术参考。
崔凤杰教授课题组近年来在真菌生物技术、真菌多糖的合成途径和关键酶系的功能解析等领域开展了一系列的研究,研究工作得到了国家自然科学基金项目(32172187,31771961,31101269)的资助,研究成果已发表在Journal of Agricultural Food and Chemistry (2019, 67 (32), 8875-8883); International Journal of Biological Macromolecules (2020, 161, 1161-1170;2020, 165, 1593-1603; 2021, 191, 996-1005);Applied Microbiology and Biotechnology ( 2022, 106, 563-578) 等期刊,获授权专利3件。
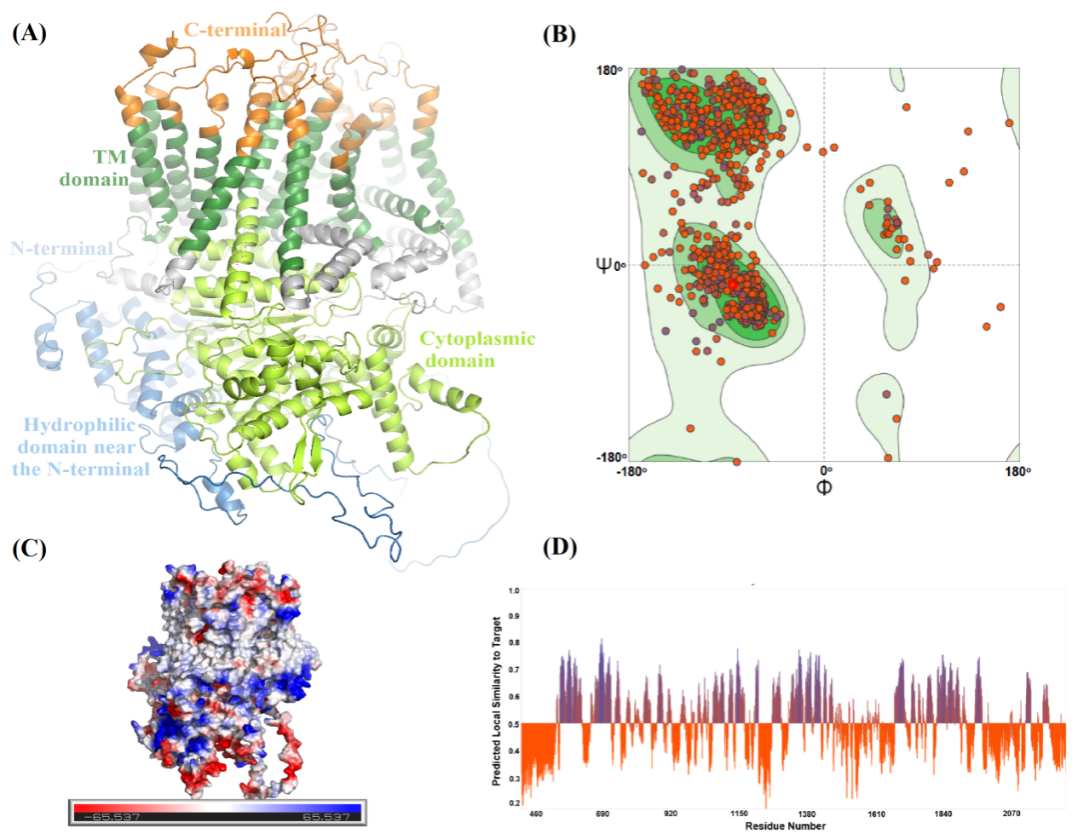
图1 AlphaFold预测的CMGLSp的3D模型
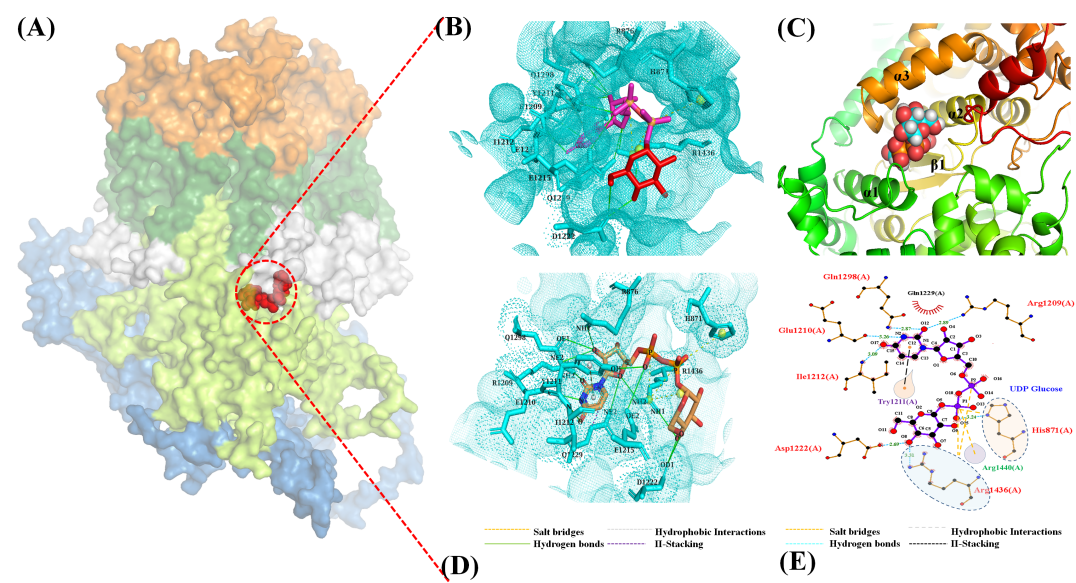
图2 CMGLS活性中心氨基酸残基与底物UDP-葡萄糖相互作用模式

